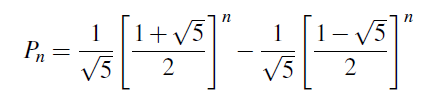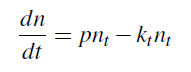Archive for the ‘Jorge Mírez’ Category
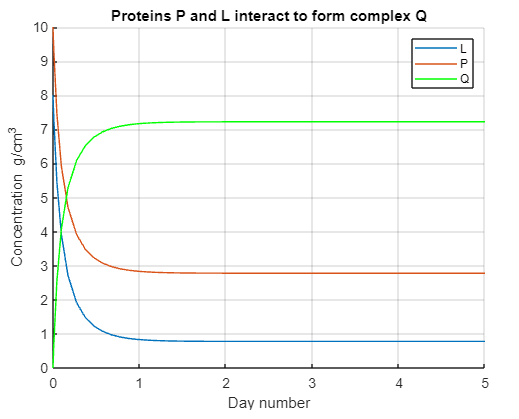
Suppose two species of proteins, P and L, interact to form a complex Q at a rate k1, while Q breaks down to its components P and L at a rate k-1. Here P , L and Q are taken as concentrations, with unit g/cm3. The reaction rate k1 is taken in unit of cm3/g · day, while the k-1 is taken in unit of 1/day. First, the differential equation has been simulate in Simulink and its results has been proccesing with MATLAB (figure has been made using MATLAB code)
Dr. Jorge Luis Mírez Tarrillo
Group of Mathematical Modeling and Numerical Simulation (GMMNS). Universidad Nacional de Ingeniería. Lima, Perú.
E-mail: jmirez@uni.edu.pe
Website Personal: https://jorgemirez2002.wixsite.com/jorgemirez
Facebook http://www.facebook.com/jorgemirezperu
Linkedin https://www.linkedin.com/in/jorge-luis-mirez-tarrillo-94918423/
Scopus ID: https://www.scopus.com/authid/detail.uri?authorId=56488109800
Google Scholar: https://scholar.google.com/citations?user=_dSpp4YAAAAJ
MATLAB Group Admin in Facebook: https://www.facebook.com/groups/Matlab.Simulink.for.All
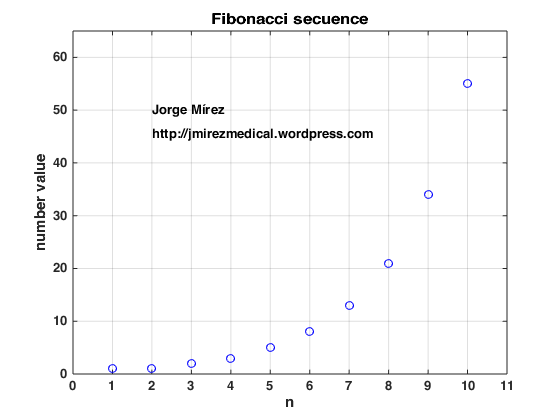
En 1202, Leonardo de Pisa, también conocido como Fibonacci, publicó un libro que popularizó en Europa el sistema numérico decimal indio que también habían adoptado los matemáticos árabes. Entre los muchos ejemplos que se dan en el libro, uno se refiere al crecimiento de una población de conejos. Es probablemente uno de los ejemplos más antiguos de un modelo matemático para la dinámica de poblaciones. La secuencia (Pn) se podía encontrar en los libros de matemáticas recreativas con el nombre de secuencia de Fibonacci. Hecho con MATLAB.
Dr. Jorge Luis Mírez Tarrillo – PERU
Página web: https://jorgemirez2002.wixsite.com/jorgemirez
Facebook http://www.facebook.com/jorgemirezperu
Linkedin https://www.linkedin.com/in/jorge-luis-mirez-tarrillo-94918423/
Scopus ID: https://www.scopus.com/authid/detail.uri?authorId=56488109800
Google Scholar: https://scholar.google.com/citations?user=_dSpp4YAAAAJ
Administrador de Grupo MATLAB en Facebook: https://www.facebook.com/groups/Matlab.Simulink.for.All
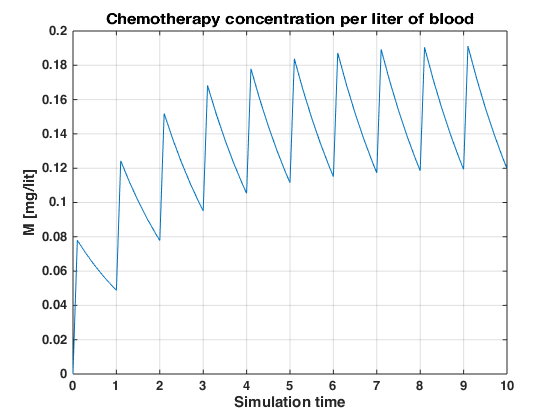
Revisando la información encuentro una ecuación diferencial de primer orden en el que se describe la evolución de la cantidad de químicos en la sangre durante una quimioterapia. La forma aserrada de la curva es porque se hace la inyección del químico en el 10 % de una unidad de medida de tiempo (usualmente es la hora) y dicho % puede variar – dado que es un modelo matemático – la tendencia hacia un valor máximo indica un equilibrio entre lo que puede ingresar y puede ser acumulado en la sangre. Hecho en MATLAB en mis investigaciones sobre cáncer.
Dr. Jorge Luis Mírez Tarrillo
Group of Mathematical Modeling and Numerical Simulation (GMMNS)
Universidad Nacional de Ingeniería, Lima, Perú.
Facebook http://www.facebook.com/jorgemirezperu
Linkedin https://www.linkedin.com/in/jorge-luis-mirez-tarrillo-94918423/
Scopus ID: https://www.scopus.com/authid/detail.uri?authorId=56488109800
Google Scholar: https://scholar.google.com/citations?user=_dSpp4YAAAAJ
E-mail: jmirez@uni.edu.pe
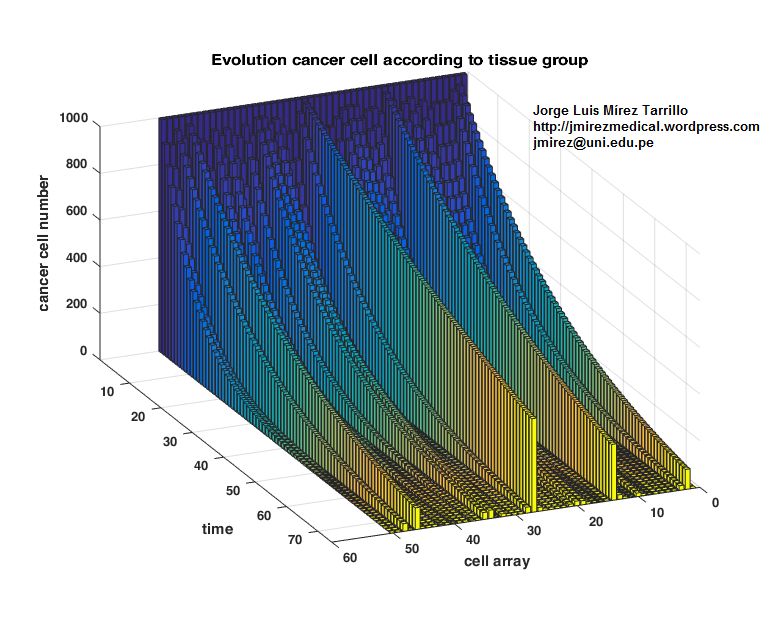
The graph shows in time line the reduction in the number of cancer cells with different proliferation rate and therapy effectiveness in a positively evolving patient… Made in MATLAB using differential equation.
Dr. Jorge Luis Mírez Tarrillo
Group of Mathematical Modeling and Numerical Simulation (GMMNS)
Universidad Nacional de Ingeniería, Lima, Perú.
Facebook http://www.facebook.com/jorgemirezperu
Linkedin https://www.linkedin.com/in/jorge-luis-mirez-tarrillo-94918423/
Scopus ID: https://www.scopus.com/authid/detail.uri?authorId=56488109800
Google Scholar: https://scholar.google.com/citations?user=_dSpp4YAAAAJ
E-mail: jmirez@uni.edu.pe
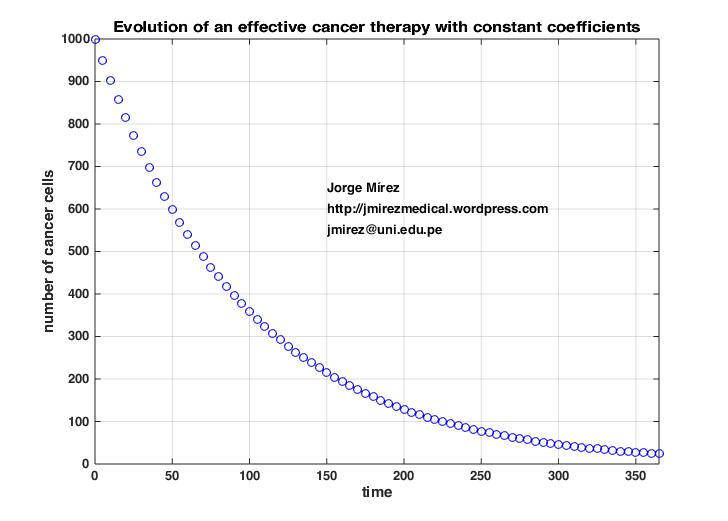
Hello readers, this figure shows the evolution of the differential equation where nt is the number of cells at time t; p is the proliferation rate of the tumor; kt is the therapy effectiveness at time t and so ktnt denotes kill rate. Made in MATLAB. I invite and welcome all support and collaboration to continue with research. Note: In this case, p and kt are constants in all simulation time.
Dr. Jorge Luis Mírez Tarrillo
Group of Mathematical Modeling and Numerical Simulation (GMMNS)
Universidad Nacional de Ingeniería, Lima, Perú.
Facebook http://www.facebook.com/jorgemirezperu
Linkedin https://www.linkedin.com/in/jorge-luis-mirez-tarrillo-94918423/
Scopus ID: https://www.scopus.com/authid/detail.uri?authorId=56488109800
Google Scholar: https://scholar.google.com/citations?user=_dSpp4YAAAAJ
E-mail: jmirez@uni.edu.pe
Hoy jueves 09 de marzo iniciamos Curso Taller de Programación en MATLAB Simulink de 7 a 10 pm (para estudiantes, tesistas, profesionales, empresas, instituciones)… No te quedes fuera, inscríbete!!. Clases virtuales vía Zoom el 09, 16, 23 y 30 Marzo. Materiales, códigos y grabaciones de clase se comparten en Drive. Costo 100 soles. Inscripciones WhatsApp 970 030 394 (Perú)


The types of available models focused on cancer biology range from general models, e.g. tumour growth, angiogenesis and signalling transduction pathway models, to specific models, e.g. behaviour in response to specific stimuli. Models of biological processes are employed for diverse research purposes, notably, inquiries into fundamental tumour dynamics and drug target prediction. Algorithms used by these models range from differential equations, use of fractal theory, stochastic approaches and more recently artificial intelligence techniques such as clustering and classification, neural networks, application of fuzzy logic, inductive/ stochastic logic programming, Bayesian networks and many more. Epidemiological models are also abundant in the literature, which are informative for the clinician in terms of modelling prognoses and diagnoses given various biological
data. The types of models presented in this report have been broadly categorised into the following classifications: Growth Models, Angiogenesis Models, Treatment Response Models and Dynamic Pathways Models. Of course not all of the models
presented here can be strictly classified into these types – in reality, all are hybrids to some degree but most comfortably fall into one of the categorisations, as portrayed by Figure. Thank you very much.

Source: Manish Patel, Sylvia Nagl. The Role of Model Integrationin Complex Systems Modelling – An Example from Cancer Biology. 2010 Springer-Verlag Berlin Heidelberg. ISBN 978-3-642-15602-1. DOI 10.1007/978-3-642-15603-8
Dr. Jorge Luis Mírez Tarrillo
Group of Mathematical Modeling and Numerical Simulation (GMMNS)
Universidad Nacional de Ingeniería, Lima, Perú.
Facebook http://www.facebook.com/jorgemirezperu
Linkedin https://www.linkedin.com/in/jorge-luis-mirez-tarrillo-94918423/
Scopus ID: https://www.scopus.com/authid/detail.uri?authorId=56488109800
Google Scholar: https://scholar.google.com/citations?user=_dSpp4YAAAAJ
E-mail: jmirez@uni.edu.pe
Una característica importante del sistema inmunitario es la capacidad de sus células constituyentes para comunicarse entre sí al encontrarse con un agente patógeno. Aunque las células del sistema inmunitario son capaces de liberar numerosas moléculas biológicamente activas con diversas funciones, dos categorías principales de proteínas (las citocinas y las quimiocinas) tienen una función muy importante en la inmunidad. Las citocinas son un grupo de proteínas que tienen efectos pleiotrópicos, como la capacidad de activar otras células, inducir la diferenciación y aumentar la actividad microbiana. Las citocinas suelen ser liberadas por las células del sistema inmunitario en respuesta a los PAMP y DAMP, y esto tiene el efecto de alterar el estado de activación y el comportamiento de otras células para impulsarlas a unirse en la lucha. Las quimiocinas también se liberan al encontrar los PAMP/DAMP y en general sirven como factores quimiotácticos, que ayudan a establecer un camino que guía a otras células del sistema inmunitario al sitio de la infección o de daño en los tejidos. Ambos tipos de proteínas mensajeras actúan al difundir fuera de las células que las secretan y al unirse a las células equipadas con los receptores adecuados en la membrana citoplasmática para recibir éstas señales.

[Fuente: Peter J. Delves, Seamus J. Martin, Dennis R. Burton, Ivan M. Roitt. «Roitt Inmunología Fundamentos» Editorial Médica Panamericana. México, 2014. ISBN: 978-607-7743-93-4]
Dr. Jorge Luis Mírez Tarrillo – PERU
Facebook http://www.facebook.com/jorgemirezperu
Linkedin https://www.linkedin.com/in/jorge-luis-mirez-tarrillo-94918423/
Scopus ID: https://www.scopus.com/authid/detail.uri?authorId=56488109800
Google Scholar: https://scholar.google.com/citations?user=_dSpp4YAAAAJ
E-mail: jmirez@uni.edu.pe
En ésta situación, las moléculas que activan el sistema inmunitario derivan de lo propio, pero no están normalmente presentes en el espacio extracelular. Éstas moléculas normalmente son secuestradas con seguridad dentro de las células sanas y sólo escapan (como si fueran «señales de peligro») cuando una célula muere a través de un modo no controlado de muerte celular, llamada necrosis; que es causada por traumatismo del tejido, quemaduras, ciertas tóxinas, y por otros estímulos no fisiológicos, y se caracteriza por la hinchazón rápida y la rotura de las membranas citoplasmáticas de las células dañadas. Esto permite la liberación de varios constituyentes celulares que normalmente no salen de las células sanas. La identidad precisa de las moléculas que actúan como señales de peligro, comúnmente llamadas patrones moleculares asociados con el peligro (DAMPS) o alarminas. En resumen: Si un agente patógeno logra eludir la detección directa por el sistema inmunitario, su presencia será relevada si provoca necrosis en el tejido que ha invadido. También existe otra forma de muerte celular, llamada apoptosis, que se utiliza para eliminar las células que han llegado al final de su vida útil natural. Las células apoptósicas no activan el sistema inmunitario debido a que las células que mueren de esta manera muestran moléculas en sus membranas citoplasmáticas que distinguen éstas células para la eliminación mediante fagocitosis antes de que se puedan romperse y liberar su contenido intracelular; de esta manera, los DAMP permanecen ocultos durante la apoptosis y estas células no activan el sistema inmunitario.

[Fuente: Peter J. Delves, Seamus J. Martin, Dennis R. Burton, Ivan M. Roitt. «Roitt Inmunología Fundamentos» Editorial Médica Panamericana. México, 2014. ISBN: 978-607-7743-93-4]
Dr. Jorge Luis Mírez Tarrillo – PERU
Facebook http://www.facebook.com/jorgemirezperu
Linkedin https://www.linkedin.com/in/jorge-luis-mirez-tarrillo-94918423/
Scopus ID: https://www.scopus.com/authid/detail.uri?authorId=56488109800
Google Scholar: https://scholar.google.com/citations?user=_dSpp4YAAAAJ
E-mail: jmirez@uni.edu.pe
La función del sistema inmunitario de los vertebrados puede ser reducida a dos funciones básicas: el reconocimiento de sustancias y organismos extraños (llamado «no propio») que han ingresado en el cuerpo y la eliminación de esos agentes por un repertorio diverso de células y moléculas que actúan en forma conjunta para eliminar la amenaza potencial. Las células y las moléculas que componen el sistema inmunitario innato están interesadas en detectar la presencia de patrones moleculares determinados que, de modo típico, se asocian a agentes infecciosos. Charlie Janeway apodó a estas moléculas como patrones moleculares con patógenos (PAMP)

[Fuente: Peter J. Delves, Seamus J. Martin, Dennis R. Burton, Ivan M. Roitt. «Roitt Inmunología Fundamentos» Editorial Médica Panamericana. México, 2014. ISBN: 978-607-7743-93-4]
Dr. Jorge Luis Mírez Tarrillo – PERU
Facebook http://www.facebook.com/jorgemirezperu
Linkedin https://www.linkedin.com/in/jorge-luis-mirez-tarrillo-94918423/
Scopus ID: https://www.scopus.com/authid/detail.uri?authorId=56488109800
Google Scholar: https://scholar.google.com/citations?user=_dSpp4YAAAAJ
E-mail: jmirez@uni.edu.pe